Participación en el X Congreso de la Sociedad Española de Biología Evolutiva (SESBE)
Durante los días 21-23 de enero de 2026, nuestro investigador predoctoral Alvaro Pita participó en el X Congreso de la Sociedad Española de Biología Evolutiva (SESBE), celebrado en el Jardin Botánico de la Universitat de València (Valencia), donde presentó los estudios genómico-poblacionales desarrollados en CONSIBERTROMICS
El resumen de nuestra comunicación se incluye a continuación:
Título: Perspectivas genómicas sobre la estructura evolutiva de los linajes de trucha común ibérica (Salmo trutta)
Autores: Álvaro Pita, Manuel Vera, David Diez del Molino, Sandra Heras, Jordi Morata, Raúl Tonda, Dorte Bekkevold, Adrián Casanova, Alba Abras, Maria Inés Roldán, Jose Luis García-Marin
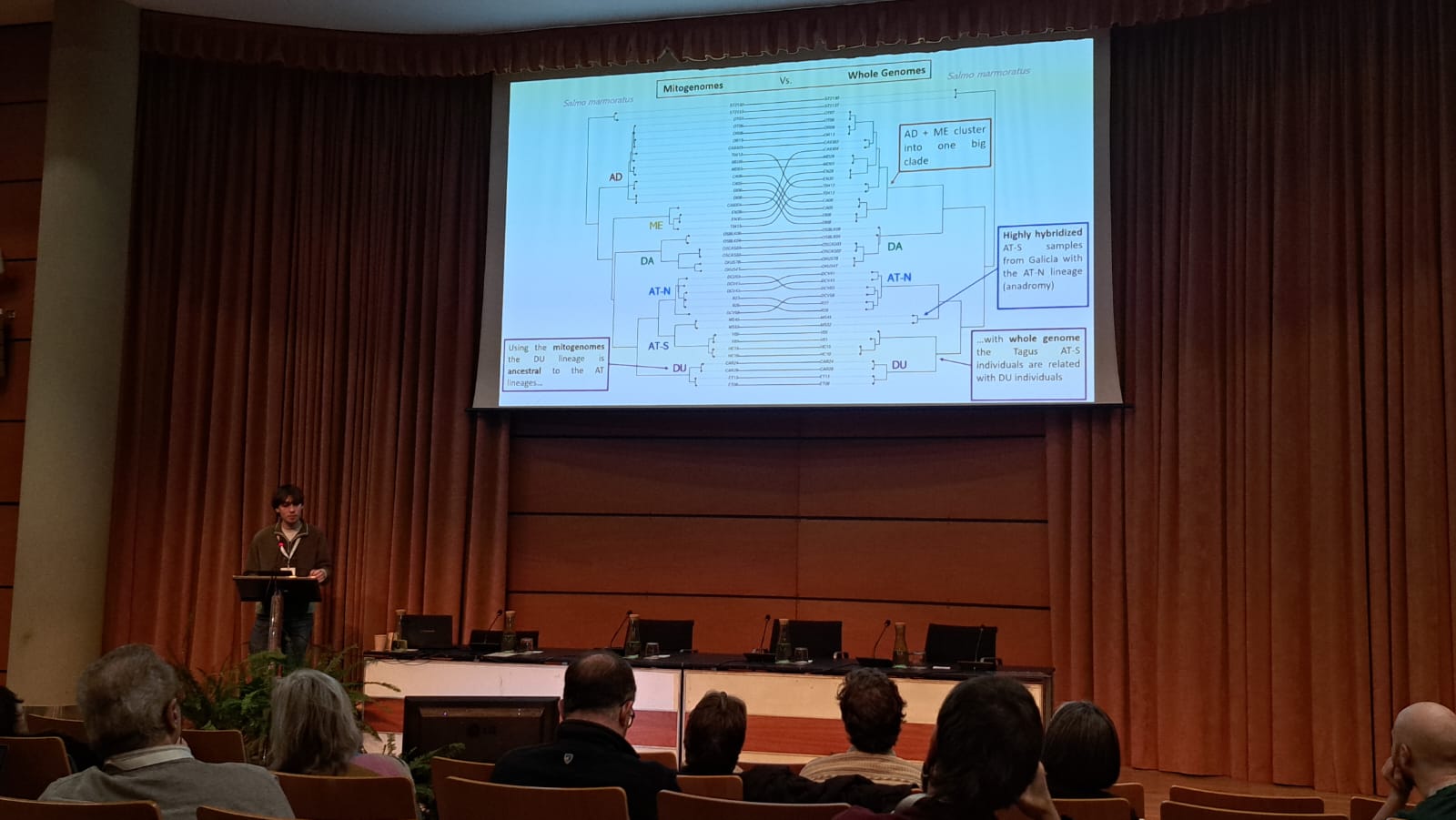
La trucha común (Salmo trutta L.) es una especie de salmónido que presenta una elevada estructuración genética y geográfica. En la Península Ibérica, la secuenciación de la región control mitocondrial identificó cuatro linajes evolutivos: Adriático (AD), Mediterráneo (ME), Atlántico (AT) y el endémico Duero (DU). Aunque la UICN restringe actualmente S. trutta a las poblaciones atlánticas, la legislación española sigue clasificando los cuatro linajes bajo una única especie. Como resultado, las poblaciones mediterráneas que engloban los linajes AD y ME, así como la trucha endémica del Duero, continúan incluidas taxonómicamente dentro de S. trutta, catalogada como de Preocupación Menor por la UICN, lo que dificulta los esfuerzos de conservación y puede favorecer la hibridación con estirpes de piscifactoría no autóctonas.
Para investigar las relaciones evolutivas entre las truchas ibéricas y otras truchas europeas, resecuenciamos los genomas de 40 individuos —26 de España y 14 de otras regiones europeas— utilizando Illumina NovaSeq 6000, empleando Salmo marmoratus como grupo externo. Las lecturas se mapearon contra el genoma de referencia de S. trutta (fSalTru1.2; GCF_901001165.2) para identificar SNPs. Los análisis filogenéticos de genomas mitocondriales, combinados con datos de GenBank, confirmaron la singularidad de los linajes ibéricos, pero también revelaron que algunas ramas incluían especies de Salmo descritas previamente. Las filogenias nucleares basadas en 4 millones de SNPs respaldaron además la profunda diferenciación entre las truchas atlánticas y mediterráneas, aunque las divergencias no se alinearon completamente con los patrones mitocondriales. Asimismo, varias poblaciones españolas mostraron claras señales de introgresión procedente de estirpes de piscifactoría, lo que pone de manifiesto el impacto genético de las prácticas de repoblación.